Correction du TD 1 avec Linux et SQLite3
Il existe pleins de manières d’extraire des données depuis des gros fichiers. Si le fichier tient en RAM ou non peut orienter le choix technologique. La correction propose plusieurs approches:
- Utilisation de Pandas
- Commande de base Linux et SQLite3
- Python pure
Préparer le fichier correctement.
Avant même de commencer à charger le fichier, il faut savoir ce qu’il
y a dedans. La commande suivante retourne la première ligne du fichier
csv, qui contient le header.
head -n1 openfoodfacts.csvcode url creator created_t created_datetime last_modified_t last_modified_datetime product_name generic_name quantity packaging packaging_tags brands brands_tags categories categories_tags categories_fr origins origins_tags manufacturing_places manufacturing_places_tags labels labels_tags labels_fr emb_codes emb_codes_tags first_packaging_code_geo cities cities_tags purchase_places stores countries countries_tags countries_fr ingredients_text allergens allergens_fr traces traces_tags traces_fr serving_size serving_quantity no_nutriments additives_n additives additives_tags additives_fr ingredients_from_palm_oil_n ingredients_from_palm_oil ingredients_from_palm_oil_tags ingredients_that_may_be_from_palm_oil_n ingredients_that_may_be_from_palm_oil ingredients_that_may_be_from_palm_oil_tags nutriscore_score nutriscore_grade nova_group pnns_groups_1 pnns_groups_2 states states_tags states_fr main_category main_category_fr image_url image_small_url image_ingredients_url image_ingredients_small_url image_nutrition_url image_nutrition_small_url energy-kj_100g energy-kcal_100g energy_100g energy-from-fat_100g fat_100g saturated-fat_100g butyric-acid_100g caproic-acid_100g caprylic-acid_100g capric-acid_100g lauric-acid_100g myristic-acid_100g palmitic-acid_100g stearic-acid_100g arachidic-acid_100g behenic-acid_100g lignoceric-acid_100g cerotic-acid_100g montanic-acid_100g melissic-acid_100g monounsaturated-fat_100g polyunsaturated-fat_100g omega-3-fat_100g alpha-linolenic-acid_100g eicosapentaenoic-acid_100g docosahexaenoic-acid_100g omega-6-fat_100g linoleic-acid_100g arachidonic-acid_100g gamma-linolenic-acid_100g dihomo-gamma-linolenic-acid_100g omega-9-fat_100g oleic-acid_100g elaidic-acid_100g gondoic-acid_100g mead-acid_100g erucic-acid_100g nervonic-acid_100g trans-fat_100g cholesterol_100g carbohydrates_100g sugars_100g sucrose_100g glucose_100g fructose_100g lactose_100g maltose_100g maltodextrins_100g starch_100g polyols_100g fiber_100g proteins_100g casein_100g serum-proteins_100g nucleotides_100g salt_100g sodium_100g alcohol_100g vitamin-a_100g beta-carotene_100g vitamin-d_100g vitamin-e_100g vitamin-k_100g vitamin-c_100g vitamin-b1_100g vitamin-b2_100g vitamin-pp_100g vitamin-b6_100g vitamin-b9_100g folates_100g vitamin-b12_100g biotin_100g pantothenic-acid_100g silica_100g bicarbonate_100g potassium_100g chloride_100g calcium_100g phosphorus_100g iron_100g magnesium_100g zinc_100g copper_100g manganese_100g fluoride_100g selenium_100g chromium_100g molybdenum_100g iodine_100g caffeine_100g taurine_100g ph_100g fruits-vegetables-nuts_100g fruits-vegetables-nuts-dried_100g fruits-vegetables-nuts-estimate_100g collagen-meat-protein-ratio_100g cocoa_100g chlorophyl_100g carbon-footprint_100g carbon-footprint-from-meat-or-fish_100g nutrition-score-fr_100g nutrition-score-uk_100g glycemic-index_100g water-hardness_100g choline_100g phylloquinone_100g beta-glucan_100g inositol_100g carnitine_100gIl n’est pas trop dure d’importer un fichier CSV dans SQLite3 à
l’aide de la ligne de commande intéractive de sqlite3.
Dans le terminal, la commande suivante va créer une base de données
sqlite3 stockée dans le fichier
openfood.db:
sqlite3 openfood.dbLes commandes suivantes sont executées dans sqlite3.
.separator "\t"
.import openfoodfacts.csv openfoodLa dernière ligne retourne des warnings
openfoodfacts.csv:57872: unescaped " character. En effet,
les champs du document csv qui contienne le symbole " sont
problématiques car il s’agit d’un caractère codant des documents csv. Le
format que nous utilisons n’utilise pas cet encodage. Pour que SQLite
lise correctement le fichier, il est donc nécessaire d’insérer le
symbôle \ avant les symbôles "
La commande suivante permet de régler le problème:
cat openfoodfacts.csv | sed 's/"/\\\\"/g' > openfoodfacts.escape.csvUne fois cette commande terminé, nous pouvons exécuté les commandes
SQL sans erreurs.
.separator "\t"
.import openfoodfacts.csv openfoodL’import du fichier peut prendre plusieurs minutes. Une fois terminé,
le fichier CSV est chargé une fois pour toute dans une base de données
SQLite. Cette dernière pèse 2.8Go contre les
2.1Go du fichier plat.
Explorer le fichier
Le nombre de lignes et le nombre de champs peuvent être obtenus par de simple
SELECT count(*) FROM openfood;1041781Extraction de lignes
L’extraction d’un sous ensemble des lignes peut s’écrire très
simplement en SQL. Par exemple pour extraire un fichier
CSV dont toutes les lignes contiennent
chocolate.
.mode csv
.output chocolate_only.csv
SELECT * FROM openfood WHERE product_name LIKE "%chocolate%";De même, il est possible de filtrer les lignes qui contiennent
chocolate ou coconuts:
.mode csv
.output chocolate_coconuts.csv
SELECT * FROM openfood WHERE product_name LIKE "%chocolate%" OR product_name LIKE "%coconuts%";Ces dernières commandes prennent chacune quelques secondes et n’utilisent que très peu de mémoire vive. Les vitesses de lectures et d’écritures sur disque sont les principaux paramètres limitant. Il faut donc faire attention avec les fichiers qui sont distants.
Extraire la liste des pays
S’il est simple de récupérer la liste des chaînes de caractères du
champs countries_fr, il est bien plus compliqué de
récupérer la liste des pays. En effet, certaines lignes contiennent des
listes de pays au format Allemagne,France.
On va utiliser ici des commandes Linux simple pour régler le
problèmes. Dans un premier temps on export la colonne
countries_fr à l’aide de la commande
.output pays_liste
SELECT countries_fr FROM openfood;Le fichier pays_liste contient donc uniquement la liste
des listes de pays. On va transformer ce fichier en liste de pays à
l’aide de la commande sed en substituant le caractère
, en un saut de ligne. Pour ça, il suffit de faire:
sed "s/,/\n/g" pays_liste > paysLe fichier pays contient donc une liste avec répétition
de tous les pays qui apparaissent dans le fichier. Pour supprimer les
répétition, on peut utiliser deux commandes:
En ligne de commande
sort pays > pays_tries
uniq -c pays_tries > pays_compteurLa commande sort trié la liste de pays par ordre
alphabétique en conservant les occurrences multiple et a stocker le
résultat dans pays_tries. La commande uniq -c
a remplacer les occurrence multiples par une seul, en comptant le
nombre. On a ainsi un fichier pays_compteur qui contient la
liste des pays et leur nombre.
Avec SQLite
Il est possible d’importer le fichier pays dans SQLite.
Pour éviter que la première ligne ne soit considérée comme l’en-tête du
fichier (comportement par défaut de la fonction .import) il
suffit de créer la table.
CREATE TABLE pays (nom TEXT);
.import pays pays
.mode csv
.output pays_compteur.csv
SELECT nom, Count(nom) FROM pays GROUP BY nom;Ces dernières lignes créent un fichier csv qui associe à
chaque pays le nombre de fois où il apparait. Si on veut n’avoir que les
pays qui apparaissent suffisamment souvent, il est possible de modifier
la requête.
CREATE TABLE pays (nom TEXT);
.import pays pays
.separator " "
.output pays_compteur.dat
SELECT * FROM
(
SELECT nom, Count(nom) as COUNT FROM pays GROUP BY nom
)
WHERE COUNT >
(
SELECT count(*)/100 FROM PAYS
)
ORDER BY COUNT;Pour dessiner un barplot de ce fichier il est possible
de le faire à l’aide d’un tableur, à l’aide de R, de Python
et de matplotlib, ou en ligne de commande à l’aide de gnuplot.
Dans ce dernier cas, il suffit d’executer un script
gnuplot avec la commande gnuplot. Par exemple,
si le fichier barplot.gnu contient les lignes
suivantes:
set terminal png size 1000,800; # Format de l'image
set output 'pays_count.png'; # Nom du fichier de sortie
set style fill solid; # Style des bars
set boxwidth 0.75;
set format y "%gK" # Format des nombre sur l'axe des abscisse
set style data histograms; # Style de plot
set xtics nomirror rotate by -45; # Style des label sur les ordonnées
plot 'pays_compteur_filter.dat' using (floor($2/1000)):xtic(1) with boxes lc rgb "dark-red" notitle;
# Indique le nom du fichier à utiliser, la valeur de l'ordonnée et de l'abscisse en fonction du contenu du fichier.
# Par défault le séparateur est un espace. Ce script produit l’image suivante
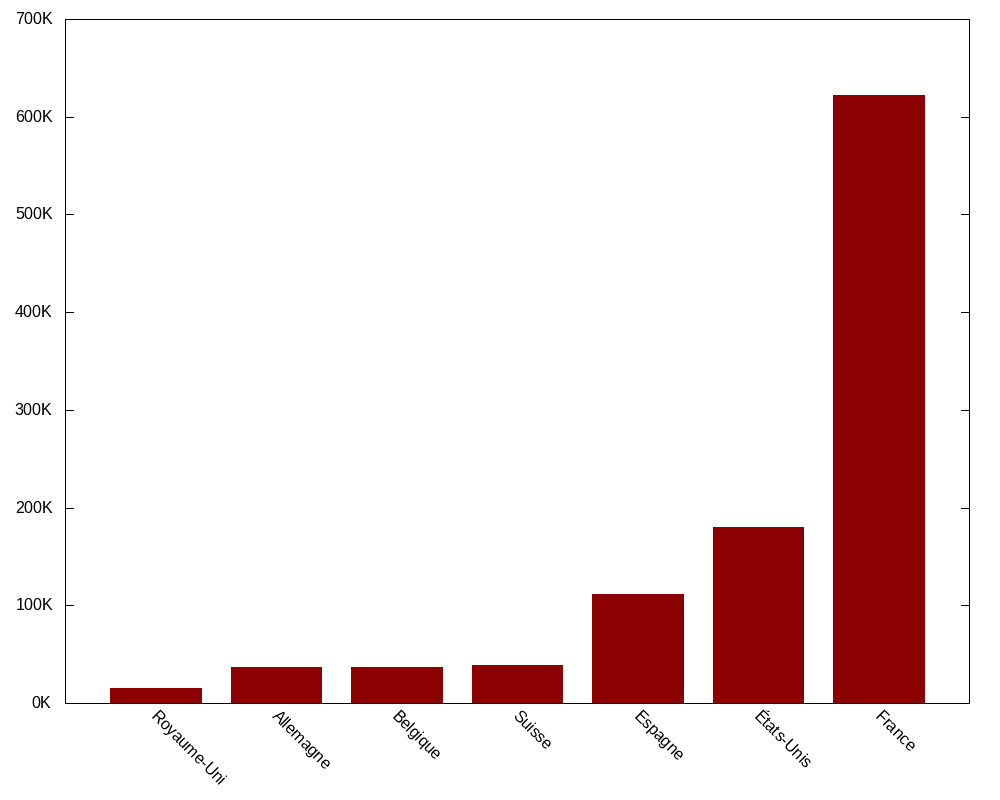
avec la commande suivante:
gnuplot barplot.gnuEnlever les champs de données vides
Nous allons maintenant comment déterminer quels sont les champs qui
contiennent peux de valeurs. Identifier ces valeurs est relativement
aisée en Python ou en utilisant un langage de programmation quelconque.
Si on se restreint à la ligne de commande et à SQLite3, c’est plus
difficile: cela nécessite de générer des requêtes SQL en
ligne de commandes.
La ligne de commande suivante va générer la liste des champs de la table.
head -n1 openfoodfacts.csv | sed 's/\t/\n/g' > fieldsva produire un fichier fields qui contient
code
url
creator
created_t
created_datetime
last_modified_t
last_modified_datetime
product_name
generic_name
quantity
packaging
packaging_tags
brands
brands_tags
...
choline_100g
phylloquinone_100g
beta-glucan_100g
inositol_100g
carnitine_100gEn utilisant le fait que sed associe le début de ligne
avec le caractère ^ et la fin de ligne avec le caractère
$, il est possible de construire une requête SQL qui compte
les champs vide pour chacune des lignes ci-dessus:
head -n1 openfoodfacts.csv | sed 's/\t/\n/g' | sed 's/^/SELECT COUNT(*) FROM openfood WHERE "/g' | sed 's/$/" = "";/g'Va produire
SELECT COUNT(*) FROM openfood WHERE "code" = "";
SELECT COUNT(*) FROM openfood WHERE "url" = "";
SELECT COUNT(*) FROM openfood WHERE "creator" = "";
SELECT COUNT(*) FROM openfood WHERE "created_t" = "";
SELECT COUNT(*) FROM openfood WHERE "created_datetime" = "";
SELECT COUNT(*) FROM openfood WHERE "last_modified_t" = "";
SELECT COUNT(*) FROM openfood WHERE "last_modified_datetime" = "";
SELECT COUNT(*) FROM openfood WHERE "product_name" = "";
SELECT COUNT(*) FROM openfood WHERE "generic_name" = "";
SELECT COUNT(*) FROM openfood WHERE "quantity" = "";
SELECT COUNT(*) FROM openfood WHERE "packaging" = "";
SELECT COUNT(*) FROM openfood WHERE "packaging_tags" = "";
SELECT COUNT(*) FROM openfood WHERE "brands" = "";
SELECT COUNT(*) FROM openfood WHERE "brands_tags" = "";
...
SELECT COUNT(*) FROM openfood WHERE "choline_100g" = "";
SELECT COUNT(*) FROM openfood WHERE "phylloquinone_100g" = "";
SELECT COUNT(*) FROM openfood WHERE "beta-glucan_100g" = "";
SELECT COUNT(*) FROM openfood WHERE "inositol_100g" = "";
SELECT COUNT(*) FROM openfood WHERE "carnitine_100g" = "";Pour exécuter ces requêtes, il suffit alors d’utiliser un tube avec
SQLite:
head -n1 openfoodfacts.csv | sed 's/\t/\n/g' | sed 's/^/SELECT COUNT(*) FROM openfood WHERE "/g' | sed 's/$/" = "";/g' | sqlite3 openfood.db > countsRemarque. Pour l’exercice faire ça en ligne de commande est intéressant, mais en situation on utilisera plutôt Python et SQLite ensemble.
Nous avons maintenant deux fichiers, un qui contient les champs et un autre qui contient les résultats de la requête. Nous allons réalisé une jointure pour les associé l’un à l’autre. Pour ce faire, nous allons utiliser le numéro de ligne comme clef de jointure.
cat -n count_fields > counts_ligne
cat -n fields > fields_ligne
join count_fields_ligne fields_ligne | sort -n > counts_fieldsCes lignes vont ainsi produire le résultat du nombre de valeur vide pour chaque champs triés.
0 code
0 countries
0 countries_fr
0 countries_tags
0 created_t
...
1041781 mead-acid_100g
1041781 melissic-acid_100g
1041781 nervonic-acid_100g
1041781 no_nutriments
1041781 water-hardness_100gNous allons garder uniquement les lignes qui ont moins de 700000 valeurs à nulles. Pour faire cela on peut utiliser une bête expression régulière. Pour identifier les champs avec beaucoup de valeurs nuls, il suffit que leur nombre s’écrivent avec 7 lettres où avec 6 lettres et débute avec le chiffre 7,8 ou 9. Cette condition s’écrit facilement avec une expression régulière. On peut en profiter pour ajouter d’autres contraintes (contient “url” dans le nom, ou contient “tag”).
Cela donne la commande suivante:
grep -E "[7-9][0-9]{5}|[0-9]{7}|url|tag" counts_fieldsComme SQLite ne supporte pas la supression de colonne,
la seule solution est de créer une nouvelle table avec seulement les
colonne qui nous intéresse. Pour ce faire, on peut utiliser l’option -v
de grep qui va retourner le résultat complémentaire.
grep -Ev "[7-9][0-9]{5}|[0-9]{7}|url|tag|_t" counts_fields | cut -f2 -d" " > important_fieldsLe fichier important_fields contient
code
countries
countries_fr
last_modified_datetime
pnns_groups_2
product_name
states
states_fr
created_datetime
creator
pnns_groups_1
energy_100g
proteins_100g
fat_100g
carbohydrates_100g
salt_100g
sodium_100g
sugars_100g
saturated-fat_100g
energy-kcal_100g
brands
additives_n
ingredients_from_palm_oil_n
categories
categories_fr
main_category
main_category_frOn va transformer ce fichier en une requête SQL qui créer une
nouvelle table. Pour ça, il faut entourer chaque nom de colonne par des
", supprimer les retours à la ligne et entouré ça des
instructions SQL. Cela donne:
cat important_fields | sed 's/^/"/g'|sed 's/$/"/g' | tr '\n' ', ' | sed 's/^/CREATE TABLE openfood2 AS SELECT /g' | sed 's/,$/ FROM openfood/g' | sqlite3 openfood.dbIl ne reste plus qu’a supprimer la table openfood et a
renommer la table openfood2 en openfood.
Éventuellement d’extraire un fichier csv propre:
DROP TABLE openfood;
ALTER TABLE openfood2 RENAME TO openfood;
.header ON
.mode csv
.output openfood.clean_sqlite.csv
SELECT * FROM openfood;Analyse de données
La suite du TP peut être fait en SQL. On souhaite extraire un tableau croisé qui associe le taux de sucre, le taux de graisse et l’énergie. On va dans un premier temps devoir lire les données comme des nombres à virgule flottante pour pouvoir faire des opération arithmétique sur leur valeur. On stock le résultat dans une table (ou dans une vue) afin d’alléger l’écriture de requêtes ultérieures.
CREATE TABLE nutrition AS
SELECT
CAST(sugars_100g as REAL) as sucre,
CAST(fat_100g as REAL) as graisse,
CAST(energy_100g AS REAL) as energie
FROM openfood
WHERE sugars_100g != "" AND
fat_100g != "" AND
energy_100g!="";Certaines lignes contiennent des valeurs incompatible avec le type de la colonne. Par exemple des colonne ou le taux de sucres est de 3700g (sur 100g). On va supprimer de la table toutes ces valeurs pour éviter de polluer les résultats de l’analyse.
DELETE FROM nutrition WHERE sucre>100 or graisse > 100;
DELETE FROM nutrition WHERE sucre < 0 or graisse < 0;
DELETE FROM nutrition WHERE sucre + graisse > 100;La précision des valeurs données n’est pas très bonne. On va donc les arrondir à la dizaine prêt.
UPDATE nutrition SET sucre = round(sucre/10)*10 and graisse = round(graisse/10)*10;On peut ainsi produire le tableau croisé qui réalise la moyenne de
l’energie en fonction du sucre et de la graisse grâce à un simple
Group By
.separator ' '
.header OFF
.output graisse_sucre_moyenne.dat
SELECT sucre, graisse, AVG(energie) FROM nutrition GROUP BY sucre, graisse order by sucre+graisse;
.output graisse_sucre_mediane.dat
SELECT sucre, graisse, G(energie) FROM nutrition GROUP BY sucre, graisse order by sucre+graisse;On a ainsi produit le tableau croisé avec des espaces comme
séparateur. On peut dessiner la heat map à l’aide d’un logiciel
quelconque ou de Pandas. Transformer la table en tableau croisé en ligne
de commande est par contre particulièrement difficile et ne sera pas
fait ici.
Compiled the: mer. 08 janv. 2025 11:50:58 CET